
- 高级人才招聘
- 中、初级专业技术人才招聘
- 管理、支撑岗位招聘
- 博士后招聘

本课题组旨在通过整合微生物多模态数据与解码大分子进化规律,建立“数据-模型-设计-构建”于一体的生物制造元件与细胞工厂创新研发体系。致力于发展从新功能元件发现、理性设计到最终高效细胞工厂构建的新方法,为生物制造提供源头核心技术与元件储备。
方向一:基因编辑工具和合成生物学催化元件的深度挖掘与设计
方向二:基于多模态人工智能模型的元件互作研究与细胞工厂构建
2012.09-2016.07 四川大学,理学学士
2016.09-2020.07 四川大学,遗传学博士
2020.11-2024.11 清华大学,博士后
2024.11至今 中国科学院微生物研究所,研究员,研究组长
2025年度 北京市科技新星
2022年度 北京生物结构前沿中心卓越学者
2020年度 清华大学结构生物学高精尖创新中心卓越学者
2019年度 国家奖学金
2017年度 唐立新奖学金
2015年度 黄乾亨奖助学金
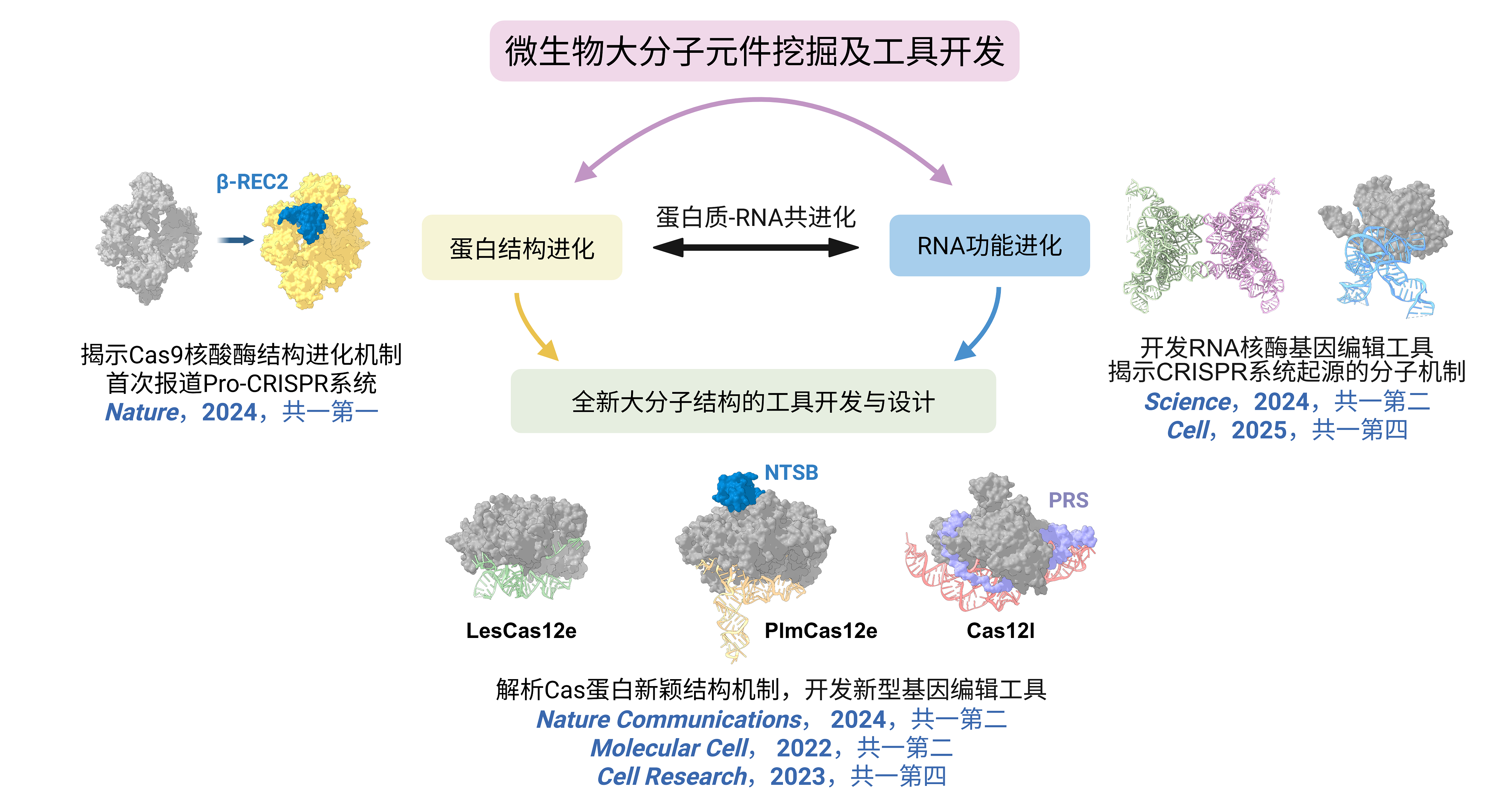
1. 基因编辑工具和合成生物学催化元件的深度挖掘与设计
聚焦生物制造中两类关键元件——基因编辑工具与生物催化元件的源头创新。我们致力于通过整合微生物多组学数据与蛋白质结构信息,系统挖掘自然界中未被充分利用的新型核酸酶系统与高性能催化酶。在此基础上,开发基于三维结构进化规律与深度学习的人工智能设计方法,实现对基因编辑工具的功能增强、特异性提升,以及对催化元件的活性优化、底物谱拓展乃至全新功能的理性设计。该研究将突破传统依赖经验试错的元件开发模式,形成“数据驱动挖掘、智能指导设计”的新范式,为微生物遗传改造与高效细胞工厂构建提供新一代底层工具与核心催化部件。
2. 基于多模态人工智能模型的元件互作研究与细胞工厂构建
致力于解决生物制造中从元件到系统的集成挑战,重点突破元件功能与细胞底盘适配性的关键瓶颈。通过整合基因组、转录组、代谢组等多模态数据,构建能够模拟微生物生理状态的“虚拟微生物”人工智能模型,系统解析代谢网络中各类元件的协同互作机制与调控规律。该研究将建立从元件功能预测到系统性能优化的全新研发范式,实现高效微生物细胞工厂的理性设计与性能提升。
代表性论文
代表性图片
申请专利
-工作人员
鞠孜敬 特别研究助理
鲁春灵 特别研究助理
王泽京 科研助理
-在读研究生
马佳月 博士生(2025- )
丁慧 博士生(2025- )
单战 硕士生(2025- )
-联培学生
袁迪 博士生(2024-)
王丰 博士生(2025-)
-本科生毕业设计
邹文瑾 本科生 (2025-)
张琨健 本科生 (2025-)
1. 科技部重点研发计划,全球微生物数据资源挖掘与利用国际合作研究,主持,2026至2028
2. 国家自然科学基金重大研究计划培育项目,细菌 RNA 演化图谱构建与耐药机制解码,主持,2026至2028
3. 中国科学院战略性先导科技专项,微生物新生命元件发掘与关键底层技术构建,研究骨干,2024至2029
4. 国家自然科学基金委员会面上项目,基于GII-B内含子的RNA核酶基因编辑工具HYER-B系统的设计和开发,主持,2025至2028
5. 北京市科技新星计划,微生物功能大分子结构进化推演及人工智能设计,主持,2025至2028
6. 国家自然科学基金委员会青年项目,新型CRISPR-Cas基因编辑工具分子 LpbCas12e的功能与机制研究,主持,2022至2024
7. 中国博士后科学基金会面上资助,新型CRISPR-CasX系统分子结构研究与优化改造,主持,2023至2024