一个细胞的“拉曼组”具有海量的信息,如何将海量的拉曼光谱数据转化为有意义、可利用的微生物学数据是一项较大的挑战。近日,微生物所付钰团队利用机器学习处理大规模、高维度复杂数据的能力,通过算法创新和优化,挖掘拉曼光谱数据中的潜在关联和非线性模式,在微生物表征研究上取得进展,发表如下两项项研究成果:
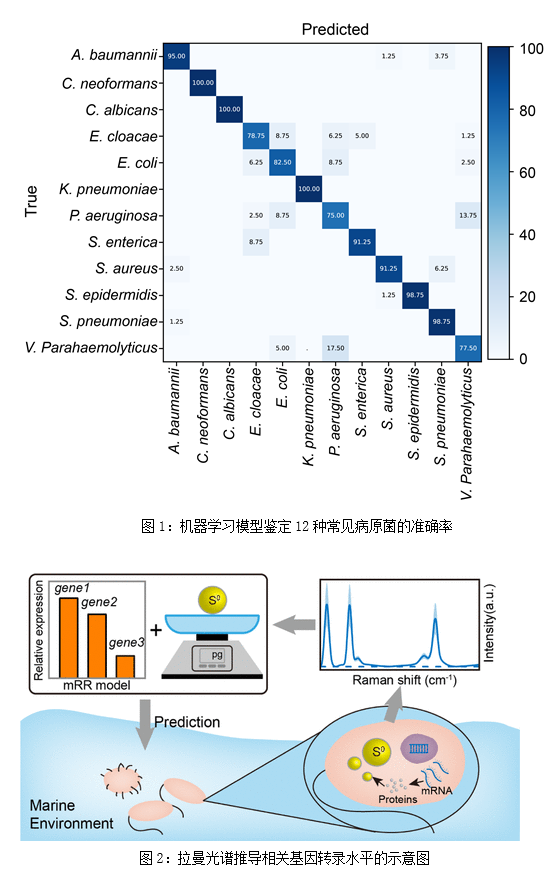
其一,将单细胞拉曼组作为微生物识别的“条形码”,通过比较一系列深度学习算法确定最佳模型,对12种常见病原微生物鉴定的平均正确率为90.73 ± 9.72%,预测鲍曼不动杆菌耐药性的准确率达到了99.92 ± 0.06%,推荐治疗抗生素类型的选择正确率为91.8%,解析了鲍曼不动杆菌耐药菌株的拉曼光谱特征,为进一步阐明耐药的分子机制奠定了基础(“Identification of pathogens and detection of antibiotic susceptibility at single-cell resolution by Raman spectroscopy combined with machine learning”, Front Microbiol, 2023)。
其二,利用数学工具建立拉曼光谱与基因转录的映射关联,实现通过拉曼光谱推算特定基因的转录水平。以海洋红树林厌氧光合紫硫细菌(Thiophaeococcus mangrovi)为例,使用拉曼光谱定量胞内硫单质,通过数学方法推导拉曼光谱、硫单质,硫球蛋白基因转录的逐一映射关联,建立mRR (mRNA and Raman)数学模型,利用拉曼光谱计算出硫球蛋白基因的转录水平。研究团队进一步在硫细菌另外两个属Thiocapsa和Thiorhodococcus中验证了该模型推导基因转录的准确性,证明了使用拉曼光谱表征基因转录的可行性,为利用无损的拉曼组在活体单细胞水平映射表征代谢组和转录组提供给了理论依据和技术创新(“Dynamically Quantifying Intracellular Elemental Sulfur and Predicting Pertinent Gene Transcription by Raman Spectroscopy in Living Cells”,Anal Chem,2023)。
微生物所付钰团队的卢维来助理研究员为论文的第一作者,付钰研究员为论文的通讯作者。本项研究工作得到了国家重点研发计划(2019YFA0905500,2021YFC2301000)、国家自然科学基金项目(52091541, 31970553, 91751103)的资助。
文章链接:
1. https://www.frontiersin.org/articles/10.3389/fmicb.2022.1076965/full
2. https://pubs.acs.org/doi/full/10.1021/acs.analchem.3c00047



