组长:丁美琪 博士 研究员

研究方向
1. 新型光遗传学生物技术的开发
2. 利用光遗传学技术调控植物生长发育
3. 解析植物免疫分子机制
研究内容和意义
1. 安全精准光遗传学工具开发
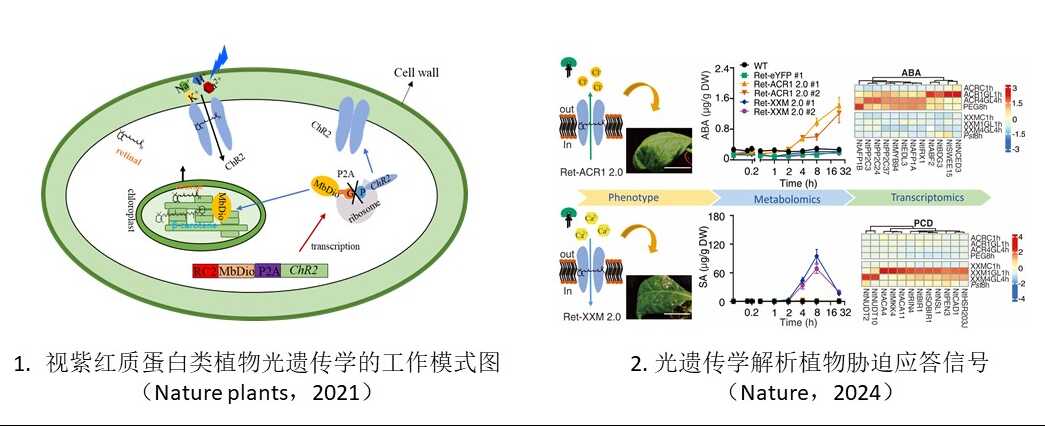
光遗传学是通过对光受体进行改造形成一类非侵害性的光控生物技术,能够实现对生命体进行可逆地精准时空调控。结合智能功能分析在微生物数据库中挖掘具有功能性的光受体蛋白并改造为具有不同特性的安全精准光遗传学工具。为微生物,植物,医学,医药化工,材料等领域提供有效的研究技术和方法。
2. 利用光遗传学探索植物生长发育机理
花粉管生长以及气孔运动对于植物生长发育至关重要。在前期的工作中我们利用光控阴离子通道进行细胞水平的调控,成功实现花粉管的生长方向的改变并调控了气孔运动。我们将进一步利用各类光控离子转运体以及光控表达原件等结合电生理技术,全光生理学技术,组学等技术进一步探索植物生长发育机理并对植物生长发育进行改造。这对高产作物研究具有积极的促进作用。
3. 植物抗病机理的研究
面对病原菌侵害时,植物会产生快速的信号响应并伴随代谢产物水平的变化与基因表达调控的改编从而形成复杂的调控网络机制。如何从复杂的调控信号网络中区分特定的信号因子是一个艰难的课题。光遗传学工具编码不同的信号因子,提供有效的研究方法进一步解析植物抗病机理。在培育新型高产抗病农作物方面具有潜在应用价值。
研究组长
丁美琪 研究员、博士生导师
电子邮件:dingmq#im.ac.cn(请将#换成@)
通讯地址:北京市朝阳区北辰西路1号院3号, H-928室,中国科学院微生物研究所
邮政编码:100101
主要学习及工作经历
2024.12至今 中国科学院微生物研究所,研究员,研究组长
2023.01-2024.07 维尔茨堡大学,博士后
2017.11-2022.12 维尔茨堡大学,生物学专业,理学博士
2014.09-2017.06 吉林大学,植物学专业,理学硕士
2010.09-2014.07 吉林大学,生物技术(植物)专业,理学学士
代表性论文
1. M. Ding, S. Gao,Non-invasive optogenetic stimulation of distinct stress responses in plants. Nature Research Briefing (2024).
2. M. Ding#, Y. Zhou#, D. Becker#, S. Yang#, M. Krischke, S. Scherzer, J. Yu-Strzelczyk, M. J. Mueller, R. Hedrich*, G. Nagel*, S. Gao*, K. R. Konrad*, Probing plant signal processing optogenetically by two channelrhodopsins. Nature 633,872–877 (2024).
3. D. Graus, K. Li, J.M. Rathje, M. Ding, M. Krischke, M.J. Muller, T.A. Cuin, K.A.S. Al-Rasheid, S. Scherzer, I. Marten, K.R. Konrad*, and R. Hedrich*, Tobacco leaf tissue rapidly detoxifies direct salt loads without activation of calcium and SOS signaling. The New phytologist 237, 217-231 (2022).
4. S. Huang#, M. Ding#, M. R. G. Roelfsema, I. Dreyer, S. Scherzer, K. A. S. Al-Rasheid, S. Gao, G. Nagel, R. Hedrich*, K. R. Konrad*, Optogenetic control of the guard cell membrane potential and stomatal movement by the light-gated anion channel GtACR1. Science advances 7, eabg4619 (2021). (Co-first author).
5. Y. Zhou#, M. Ding#, S. Gao#*, J. Yu-Strzelczyk#, M. Krischke, X. Duan, J. Leide, M. Riederer, M. J. Mueller, R. Hedrich, K. R. Konrad*, G. Nagel*, Optogenetic control of plant growth by a microbial rhodopsin. Nature plants 7, 144-151 (2021). (Co-first author).
6. Y. Zhou, M. Ding, G. Nagel, K. R. Konrad, S. Gao*, Advances and prospects of rhodopsin-based optogenetics in plant research. Plant physiology 187, 572-589 (2021).
7. Y. Zhou, M. Ding, X. Duan, K. R. Konrad, G. Nagel, S. Gao*, Extending the Anion Channelrhodopsin-Based Toolbox for Plant Optogenetics. Membranes 11, (2021).
8. M. Ding#, H. Dong#, Y. Xue, S. Su, Y. Wu, S. Li, H. Liu, H. Li, J. Han, X. Shan*, Y. Yuan*, Transcriptomic analysis reveals somatic embryogenesis-associated signaling pathways and gene expression regulation in maize (Zea mays L.). Plant molecular biology 104, 647-663 (2020).
9. B. Liu, S. Su, Y. Wu, Y. Li, X. Shan, S. Li, H. Liu, H. Dong, M. Ding, J. Han, Y. Yuan, Histological and transcript analyses of intact somatic embryos in an elite maize (Zea mays L.) inbred line Y423. Plant Physiol Bioch 92, 81-91 (2015).
代表性图片