近日,中国科学院微生物研究所吴边和崔颖璐团队在The Innovation上发表题为“Structure-based self-supervised learning enables ultrafast protein stability prediction upon mutation”的研究论文。该研究开发了一种基于结构自监督学习的深度学习框架Pythia,可以快速预测蛋白质突变的稳定性变化(ΔΔG)。该研究不仅揭示了蛋白质三维结构蕴含的进化语义信息与热力学稳定性之间的深层关联,还通过开源代码(GitHub链接)与Web服务器技术服务体系,为蛋白质工程领域提供了便捷的研究工具。蛋白质突变效应预测是解码分子进化机制、优化蛋白质工程改造的关键物理量。然而,传统预测方法面临两大挑战:一是基于物理力场的计算方法(如自由能微扰)计算复杂度高,难以满足大规模筛选需求;二是依赖于实验数据的监督学习方法易受训练集偏差影响,泛化能力受限。针对这些问题,研究团队提出了基于三维结构自监督学习的深度学习框架Pythia。该框架融合了图神经网络与注意力机制,可直接从蛋白质结构中学习氨基酸之间的相互作用。这种"零监督"预训练策略突破了传统方法对标记数据的依赖性,成功捕获了蛋白质折叠过程中隐藏的物理化学约束规律。
相比于传统力场函数方法,Pythia单点突变ΔΔG预测速度提升5个数量级,单核计算速度达到约50,000个突变/分钟。在标准测试集S2648上,Pythia的Spearman相关系数为0.616,Pearson相关系数为0.598,性能优于对比的所有现有模型。这一突破性进展为大规模扫描蛋白质序列空间提供了高效工具。在湿实验验证中,Pythia在不依赖专家筛选的情况下,成功率比基于能量函数的方法提高了一倍,充分证明了其在实际应用中的可靠性。未来,该框架可进一步拓展至蛋白质-蛋白质相互作用预测、多突变协同效应分析等复杂场景,助力推动计算驱动蛋白质设计范式的转型升级。
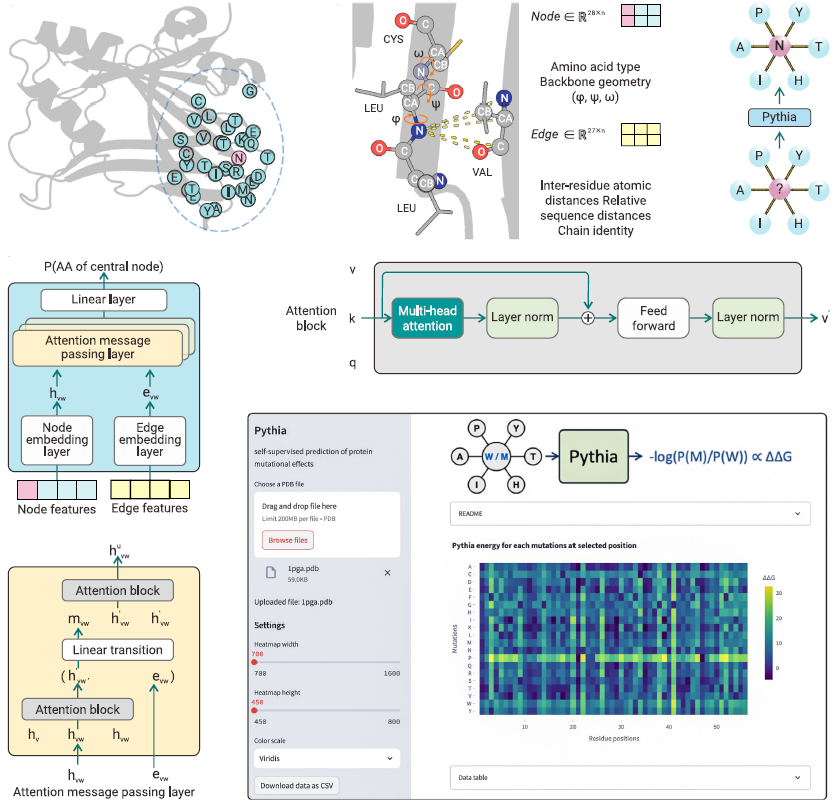
图1. Pythia模型框架
中国科学院微生物研究所博士研究生孙瑨原为本文第一作者,微生物所吴边研究员和崔颖璐项目研究员为本文共同通讯作者。本研究工作得到了国家重点研发计划、国家自然科学基金、中国科学院战略性先导科技专项和中国科学院青年创新促进会的支持。
论文链接:https://doi.org/10.1016/j.xinn.2024.100750




