感染免疫表观遗传调控的分子机制研究团组
组长:杨娜 博士、研究员

研究方向
感染免疫表观遗传调控的分子机制研究,聚焦病原蛋白与宿主表观遗传因子相互调节的结构机理和化学干预研究。
研究内容和意义
杨娜博士多年从事表观遗传调控的结构机理及化学干预研究。针对新型组蛋白修饰酶复合体、DNA甲基化因子和核酸结合蛋白开展了酶活性调控、底物识别特异性的结构机理研究,以期阐明这些表观遗传修饰建立、解读和调控下游基因表达,影响生物免疫和引发疾病的分子机制。加入微生物研究所后工作聚焦感染免疫表观遗传调控的分子机制研究,开展病原蛋白与宿主表观因子相互调节的结构药理学研究,开展致病分子机理、药物靶点论证以及结构和AI指导的理性药物设计研究,促进预防和治疗致病性感染有效疗法的开发。
具体的研究方向包括:
1.病原与宿主基因表达交互调控的表观机制研究。
2.病原蛋白调节宿主染色质高级结构影响基因表达的分子机理研究。
3.表观遗传因子参与T细胞发育的分子机制以及化学干预T细胞耗竭的结构药理学研究。
研究组长
杨娜
电子邮件:yangna#im.ac.cn(请将#换成@)
通讯地址:北京市朝阳区北辰西路1号院3号,中国科学院微生物研究所
邮政编码:100101
主要学习及工作经历
1996.09-2000.07 北京大学,生命科学学院,学士
2000.09-2005.07 中国科学院生物物理研究所,生物化学与分子生物学专业,博士
2005.07-2006.03中国科学院生物物理研究所,研究助理
2006.04-2008.09 北京大学深圳研究生院,博士后
2008.10-2013.11 中国科学院生物物理研究所,生物大分子国家重点实验室,副研究员
2013.12-2017.04 中国科学院生物物理研究所,生物大分子国家重点实验室,研究员
2015.10-2017.04 中国科学院大学,生命科学学院,岗位教授、博导
2017.04-2024.11 南开大学,药学院、药物化学生物学全国重点实验室,教授
2024.11-至今 中国科学院微生物研究所,病原微生物与免疫学重点实验室,研究员,研究组长
获奖情况
2019 第十五届天津青年科技奖
2019 天津市引进领军人才创新类
2018 北京市科学技术奖二等奖(第五完成人)
2017 天津市杰出青年科学基金
2016 国家优秀青年科学基金
2015 中国科学院青年创新促进会优秀会员
2011 中国科学院卢嘉锡青年人才奖
2011 中国科学院青年创新促进会会员
2006 全国博士后科学基金资助一等奖
2005 中国科学院院长奖优秀奖
2004 中国科学院生物物理研究所所长奖学金一等奖
2001 中国科学院优秀研究生奖学金
学术服务
2021-今 中国生物物理学会分子生物物理分会,副理事长
2015-2021 中国生物物理学会分子生物物理分会,秘书长
2019-今 中国遗传学会表观遗传学分会,委员,秘书处负责人
2016-2021 中国晶体学会第六届理事会,理事
2015-2019 中国科学院青年联合会第四届委员会委员
2014-2023 国际晶体学联合会(IUCr),大分子专业委员会顾问委员
2018-2019 世界经济论坛青年科学家
代表性论文
1. Shi, F.D.#, Zhang, K.#, Cheng, Q.X., Che, S.Y., Zhi, S.X., Yu, Z.Y., Liu, F., Duan, F.F., Wang, Y.M.* and Yang, N.* (2024) Molecular mechanism governing RNA-binding property of mammalian TRIM71 protein. Science Bulletin, Vol. 69, 72–81.
2. Yang, J.#, Dan, J.M.#, Zhao, N.N., Liu, L.L., Wang, H.S., Liu, Q.Q., Wang, L.L., Li, J., Wu, Y.W., Chen, F.L., Fu, W.L., Liu, F., Lin, M.Q., Zhang, W.Y., Chen, F.Q., Liu, X.Q., Lv, X.Y., Chen, Q., Wu, X.D., Niu, Y.Y.*, Yang, N.*, Zhu, Y.S.*, Long, J.F.*, Liu, L.* (2024) Zscan4 mediates ubiquitination and degradation of co-repressor complex to promote chromatin accessibility in 2C-like cells. Proc. Natl. Acad. Sci. USA, in press.
3. Liu, F., Wang, J.*, Xu, R.M.* and Yang, N.* (2023) Energy landscape quantifications of histone H3.3 recognition by chaperon DAXX reveal an uncoupled binding specificity and affinity. Physical Chemistry Chemical Physics, Vol. 25, 27981–27993.
4. Liu, C.P.#, Yu, Z.#, Xiong, J.#, Hu, J.#, Song, A.#, Ding, D., Yu, C., Yang, N., Wang, M., Yu, J., Hou, P., Zeng, K., Li, Z., Zhang, Z., Zhang, X., Li, W., Zhang, Z., Zhu, B.*, Li, G.* and Xu, R.M.* (2023) Structural insights into histone binding and nucleosome assembly by chromatin assembly factor-1. Science, Vol. 381, eadd8673.
5. Liu, F.#, Pang, N.N.#, Xu, R.M. and Yang, N.* (2023) Mechanism and design of allosteric activators of SIRT1. Protein & Cell, Vol. 14, 387-392.
6. Sun, J.X., Liu, F., Yuan, L.X., Pang, N.N., Zhu, B. and Yang, N.* (2023) Mechanism studies of the activation of DNA methyltransferase DNMT1 triggered by histone H3 ubiquitination, revealed by multi-scale molecular dynamics simulations. Science China Life Sciences, Vol. 66, 313-323.
7. Pang, N.N.#, Sun, J.X.#, Che, S.Y. and Yang, N.* (2022) Structural study of fungus-specific histone deacetylase Hos3 and insight into developing selective inhibitors with antifungal activity. Journal of Biological Chemistry, Vol. 298, 102068.
8. Ma, S.#, Zhang, J.Y.#, Guo, Q.S., Cao, C., Bao, K.W., Liu, L., Chen, D.G., Liu, Z., Yang, J., Yang, N.*, Yao, Z.* and Shi, L.* (2022) Disrupting PHF8-TOPBP1 connection elicits a breast tumor-specific vulnerability to chemotherapeutics. Cancer Letters, Vol. 530, 29-44.
9. Ma, S.#, Cao, C.#, Che, S.Y.#, Wang, Y.J.#, Su, D.X.#, et al., Yao, Z.*, Yang, N.* and Shi, L.* (2021) PHF8-promoted TOPBP1 demethylation drives ATR activation and preserves genome stability. Science Advances, Vol. 7, eabf7684.
10. Xu, X.#, Wang, M.Z.#, Sun, J.X.#, Yu, Z.Y.#, Li, G.H., Yang, N.* and Xu, R.M.* (2021) Structure specific DNA recognition by the SLX1-SLX4 endonuclease complex. Nucleic Acid Research, Vol. 49, 7740-7752.
11. Sun, J.X.#, Li, Z.B#. and Yang, N.* (2021) Mechanism of the conformational change of the protein methyltransferase SMYD3: a molecular dynamics simulation study. International Journal of Molecular Sciences, Vol. 22, 7185 (1-21).
12. Liu, F. and Yang, N.* (2020) Multiscale landscape of molecular mechanism of SIRT1 activation by STACs. Physical Chemistry Chemical Physics, Vol. 22, 826-837.
13. Song, X.S.#, Yang, L.L.#, Wang, M.Z., Gu, Y., Ye, B.Q., Fan, Z.S., Xu, R.M.* and Yang, N.* (2019) A higher-order configuration of the heterodimeric DOT1L-AF10 coiled-coil domains potentiates their leukemogenenic activity. Proc. Natl. Acad. Sci. USA, Vol. 116, 19917-19923.
14. Sun, J.X., Shi, F.D. and Yang, N.* (2019) Exploration of the substrate preference of lysine methyltransferase SMYD3 by molecular dynamics simulations. ACS Omega, Vol. 4, 19573-19581.
15. Li, Y.#, Duan, F.F.#, Zhao, Y.T., Gu, K.L., Liao, L.Q., Su, H.B., Hao, J., Zhang, K., Yang, N. and Wang Y.M.* (2019) A TRIM71 binding long noncoding RNA Trincr1 represses FGF/ERK signaling in embryonic stem cells. Nature Communications, Vol. 10, 1368.
16. Zhang, L.#, Serra-Cardona, A.#, Zhou, H., Wang, N., Yang, N., Zhang, Z.* and Xu, R.M.* (2018) Multisite substrate recognition in Asf1-dependent acetylation of histone H3 K56 by Rtt109. Cell, Vol. 174, 818-830.
17. Fu, W.Q.#, Liu, N.#, Qiao, Q.#, Wang, M., Min, J.R., Zhu, B.*, Xu, R.M.* and Yang, N.* (2016) Structural Basis for Substrate Preference of SMYD3, A SET Domain-containing Protein Lysine Methyltransferase. Journal of Biological Chemistry, Vol. 291, 9173-9180.
18. Fang, D.#, Gan, H.#, Lee, J.H.#, Han, J.#, Wang, Z.#, Riester, S.M., Jin, L., Chen, J., Zhou, H., Wang, J., Zhang, H., Yang, N., Bradley, E.W., Ho, T.H., Rubin, B.P., Bridge, J.A., Thibodeau, S.N., Ordog, T., Chen, Y., van Wijnen, A.J., Oliveira, A.M., Xu, R.M., Westendorf, J.J. and Zhang, Z.* (2016) The histone H3.3K36M mutation reprograms the epigenome of chondroblastomas. Science, Vol. 29, 1316-1325.
19. Cao, D.F., Wang, M., Qiu, X.Y., Liu, D.X., Jiang, H.L., Yang, N.* and Xu, R.M.* (2015) Structural basis for allosteric, substrate-dependent stimulation of SIRT1 activity by resveratrol. Genes & Development, Vol. 29, 1316-1325.
20. Yang, D.X.#, Fang, Q.L.#, Wang, M.#, Ren, R., Wang, H., He, M., Sun, Y.W., Yang, N.* and Xu, R.M.* (2013) Nα-acetylated Sir3 stabilizes the conformation of a nucleosome-binding loop in the BAH domain. Nature Structural & Molecular Biology, Vol. 20, 1116-1118.
代表性图片
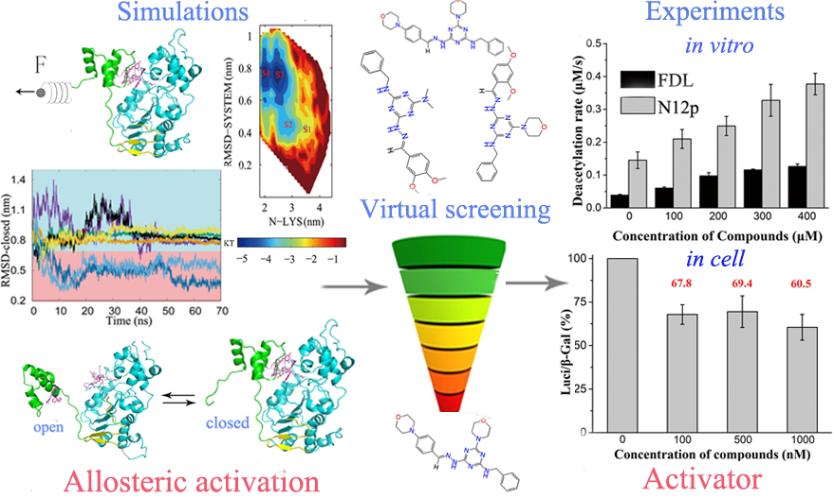
申请专利
1. 杨娜, 董利鹏, 尚鲁庆, 梁笑, 李晶晶, 沈辰. 组蛋白去甲基化酶LSD1的酶活检测体系、检测方法及应用,授权号:ZL202110333438.1
2. 杨娜, 刘飞, 庞宁宁. 一种去乙酰化酶SIRT1变构激动剂及其应用,授权号:ZL202111042375.0
3. 石磊、杨娜、姚智、马帅、曹程、车世友. 一种多肽及其在制备治疗乳腺癌药物中的应用,授权号:ZL202011247065.8
4. 杨娜,苏波,孙霁雪,袁龙啸,于海燕,杨泽坤. 一种DNA甲基转移酶DNMT1的双底物抑制剂及其应用, 授权号:ZL202310074289.0
5. 杨娜, 董利鹏, 李汀. 一种蛋白层析纯化系统,授权号:ZL201920792918.2
6. 杨娜, 武桐,黄佳欣,高闯闯,李汀. 一种蛋白质层析纯化恒温自动控制系统,授权号:ZL202320780808.0
1.《承续的魅力-令人着迷的表观遗传学》
科学出版社, 2018, ISBN 978-7-03-059640-6.
2.《Handbook of Proteolytic Enzymes》
Elsevier Academic Press, 2013, ISBN 978-0-12-382219-2.
3.《现代蛋白质工程实验指南》译
科学出版社, 2011, ISBN 978-7-03-030122-2.




